- Este post fue publicado originalmente en el blog de Escuela de Datos Vivos, para acceder al post original consultar el siguiente enlace Datos geoespaciales, futbol y delitos con Tidymodels 📦 (Workflows Parte 2/2).
Resumen 📖
📌 En el artículo anterior (Workflows Parte 1/2), se mostró de manera introductoria cómo crear modelos, recetas y workflows dentro del ecosistema tidymodels 🙌. En este post se mostrarán aplicaciones más avanzadas de los workflows 🔁 y recetas 🍳, específicamente a través de los siguientes casos de uso:
Discretización de localizaciones 📐(lat/long) con modelos predictivos.
Pivot de puntos en el mapa 🗺 (estadios ⚽ de Boca y River)
Remuestreo espacial 🧪
👉🏻 Se continuará utilizando el conjunto de datos referente a los delitos ocurridos en la ciudad de Buenos Aires, almacenado en {sknifedatar} 📦.
📌Librerías 📚
Discretización de variables con modelos predictivos
❗En el siguiente gráfico se observan las esquinas de CABA, además se agrega el número de delitos registrados durante los últimos 12 meses (situándonos en diciembre del 2019) en sus cercanías.
esquinas_plot <- sknifedatar::data_crime_clime %>%
filter(pliegue == 13) %>%
mutate(delitos_last_12 = ifelse(delitos_last_12 > 200 ,200,delitos_last_12))
ggplot(esquinas_plot, aes(x = long, y = lat), color = delitos_last_12) +
geom_point(aes(colour = delitos_last_12)) +
scale_color_gradient(low = "yellow", high = "red") +
theme_minimal()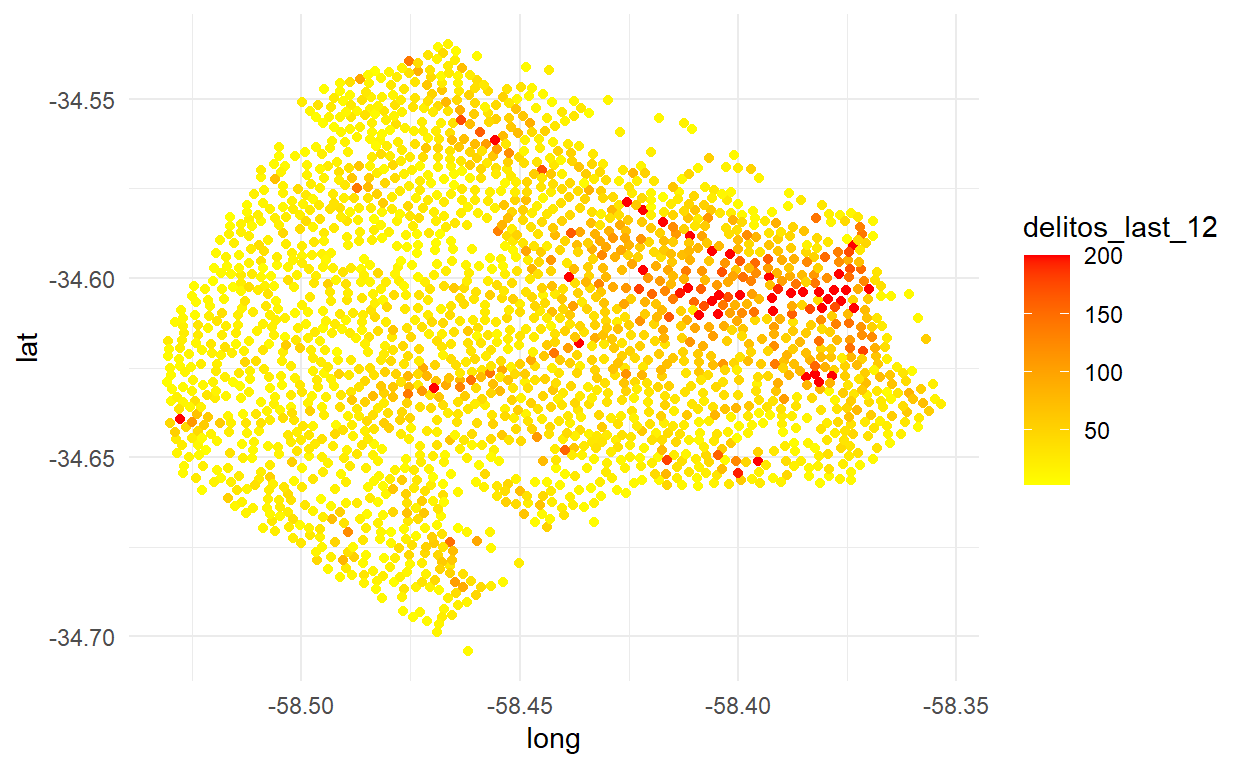
🔎 Si se busca segmentar o discretizar las esquinas en función de la relación entre la longitud y la ocurrencia de delitos 🚨, podríamos hacerlo a través de un modelo predictivo. Por ejemplo:
Receta_xgb <- recipe(esquinas_plot) %>%
step_discretize_xgb(long, outcome = "delitos_last_12") %>%
prep()👉🏻 La función step_discretize_xgb() realiza una discretización a través de un modelo de Extreme Gradient Boosting (XGBoost) 🤖, este requiere que se especifique la(s) variable a discretizar, además de la variable dependiente para ajustar el modelo.
📌 El ajuste se realiza con los parámetros por defecto, para aplicaciones más avanzadas se pueden optimizar los hiperparámetros del XGB para la discretización 💡, solo se debe agregar la función tune() a la receta y realizar el ajuste, este tópico está fuera del alcance de este post. A continuación, observamos los cortes generados.
[1] -58.49194 -58.47963 -58.46792 -58.42710 -58.40813❗Estos valores representan los puntos de longitud óptimos para segmentar las esquinas en función de los crímenes registrados. Horneando la receta sobre los datos, se muestra la variable long discretizada para cada esquina.
# A tibble: 6 x 2
id long
<fct> <fct>
1 esquina_1 [-58.48,-58.47)
2 esquina_10 [-58.41, Inf]
3 esquina_100 [-58.49,-58.48)
4 esquina_1001 [-Inf,-58.49)
5 esquina_1002 [-Inf,-58.49)
6 esquina_1003 [-Inf,-58.49) 📌Para entender el objetivo de la discretización, se muestra el mismo mapa de las esquinas de CABA pera agregando la discretización por zonas en función de la longitud y ocurrencia delictiva en los últimos 12 meses.
ggplot(esquinas_plot, aes(x = long, y = lat), color = delitos_last_12) +
geom_vline(xintercept = cortes, col = "blue", alpha = 0.7) +
geom_point(aes(colour = delitos_last_12), alpha = 0.5) +
scale_color_gradient(low = "yellow", high = "red") +
theme_bw()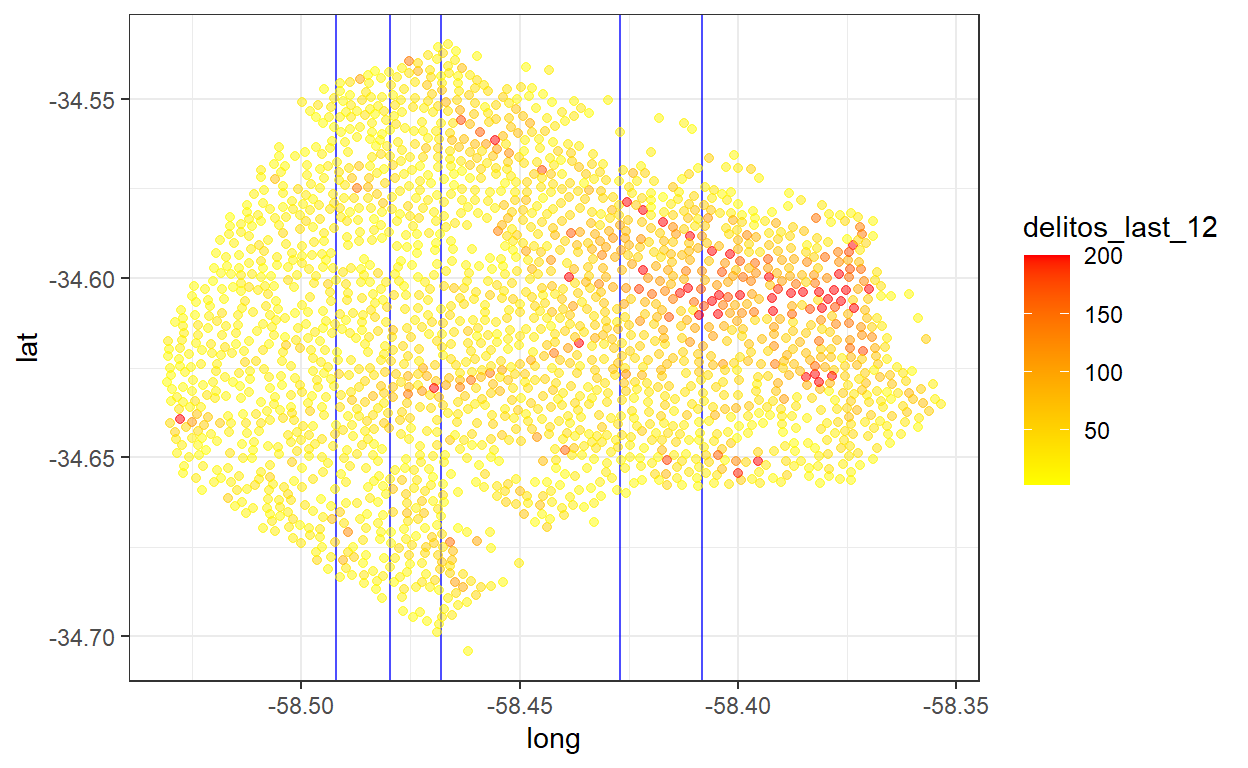
⚠️ ¿Tiene algún fundamento la implementación de esta discretización?
No, simplemente sirve para mostrar una de las tantas cosas que podemos hacer ✅. Sin embargo, en el caso de requerir realizar segmentaciones en función a otras variables, esta puede ser una opción.
👉🏻 Aunque en esta oportunidad solo se discretizo la longitud, se puede hacer también con la latitud, simplemente agregando otra step_ function.
Aplicación de la discretización sobre nuevas esquinas
🔎Para hacer un caso de uso “real”, se crearán 10 esquinas artificiales 🧙🏻 con la siguiente sintaxis (estamos obviando toda la complejidad y teoría detrás de la generación de datos sintéticos, es un simple ejemplo).
# A tibble: 10 x 55
id pliegue delitos_last_ye~ delitos_last_12 delitos_last_6
<fct> <int> <dbl> <dbl> <dbl>
1 esquina_281 13 3 26 12
2 esquina_21~ 13 4 44 20
3 esquina_403 13 1 17 8
4 esquina_17~ 13 1 26 16
5 esquina_16~ 13 0 13 5
6 esquina_617 13 0 8 3
7 esquina_774 13 13 79 40
8 esquina_17~ 13 4 36 17
9 esquina_21~ 13 0 28 16
10 esquina_22~ 13 0 20 11❗Aplicamos la receta sobre las esquinas sintéticas.
# A tibble: 10 x 2
id long
<fct> <fct>
1 esquina_281 [-Inf,-58.49)
2 esquina_2198 [-58.41, Inf]
3 esquina_403 [-58.48,-58.47)
4 esquina_1718 [-58.47,-58.43)
5 esquina_1659 [-58.48,-58.47)
6 esquina_617 [-58.41, Inf]
7 esquina_774 [-58.48,-58.47)
8 esquina_1764 [-58.43,-58.41)
9 esquina_2158 [-58.47,-58.43)
10 esquina_2284 [-Inf,-58.49) ✅ La columna longitud de las 10 esquinas fue discretizada en función de los rangos entrenados por la receta.
Pivote con puntos en el mapa 📐
📌Para fines ilustrativos se selecciona la localización del estadio de Boca Juniors y River Plate.
estadios <- data.frame(lugar = c("Boca","River"),
long = c(-58.364521, -58.449715),
lat = c(-34.635706, -34.545586))
leaflet() %>%
addTiles() %>%
addMarkers(data = estadios,
lng = ~long, lat = ~lat)¿Cuál es el objetivo? 🤔
🔘 Se tomarán estos dos puntos en el mapa como pivote para calcular la distancia entre las esquinas y estos dos puntos.
¿Por qué?
👉🏻 Simplemente para mostrar otra herramienta de utilidad dentro de las recetas. A continuación, se aplica la función step_geodist().
Receta_pivote <- recipe(esquinas_plot) %>%
step_geodist(lat = lat, lon = long, log = FALSE,
ref_lat = -34.635706, ref_lon = -58.364521, name = "dist_boca") %>%
step_geodist(lat = lat, lon = long, log = FALSE,
ref_lat = -34.545586, ref_lon = -58.449715, name = "dist_river") %>%
prep()🔎 La función requiere especificar cómo están nombradas lat y long en el dataset, el punto de latitud y longitud del estadio y finalmente el nombre de la variable a generar. A continuación, se hornea 🍳 la receta y se observan los resultados.
bake(Receta_pivote, new_data = esquinas_plot,
c(id, long, lat ,dist_boca,dist_river)) %>% #Esta linea es solo para seleccionar columnas
print(n_extra = 0)# A tibble: 2,023 x 5
id long lat dist_boca dist_river
<fct> <dbl> <dbl> <dbl> <dbl>
1 esquina_1 -58.5 -34.5 14753. 2446.
2 esquina_10 -58.4 -34.6 3797. 9687.
3 esquina_100 -58.5 -34.6 13102. 5433.
4 esquina_1001 -58.5 -34.6 15012. 5551.
5 esquina_1002 -58.5 -34.6 14373. 5146.
6 esquina_1003 -58.5 -34.6 14785. 7312.
7 esquina_1004 -58.5 -34.6 14674. 7092.
8 esquina_1005 -58.5 -34.6 14703. 7804.
9 esquina_1006 -58.5 -34.6 14694. 7470.
10 esquina_1007 -58.5 -34.6 14184. 7439.
# ... with 2,013 more rowsUniendo todo en un workflow único 🏆
📌Con el objetivo de mostrar el abanico de herramientas que ofrecen las recetas, se agregan de manera aleatoria algunos valores nulos en las variables de estaciones de servicio y locales gastronómicos. Posteriormente, en la receta se aplicará un método de imputación.
data_bonus <- sknifedatar::insert_na(.dataset = sknifedatar::data_crime_clime,
columns = c("gasolina","gastronomica"),
.p = 0.99) - Creación del modelo y partición ➗ de datos.
Modelo <- rand_forest() %>%
set_engine("ranger") %>%
set_mode("regression")
set.seed(123)
splits <- initial_split(data_bonus, prop = 0.8, strata = delitos)
splits<Analysis/Assess/Total>
<21038/5261/26299>- 🥘 Receta: Se utilizará como base la primera receta construida y agregaremos algunos pasos extra de preprocesamiento, incluyendo la discretización de la longitud y el pivote con los estadios de fútbol.
✔ Se eliminan las columnas id y pliegues. ❌
✔ Las variables gasolina y gastronomica presentan valores nulos, estos serán imputados a través de la mediana (no es el más indicado, se aplica por simplicidad, también están disponibles las imputación por knn y bagging).
✔ Se utiliza la ubicación del estadio de Boca Juniors para medir la distancia con las esquinas. 📍
✔ Se utiliza la ubicación del estadio de River Plate para medir la distancia con las esquinas. 📍
✔ Se discretiza la variable longitud, mediante un XGB en función de los delitos ocurridos en los últimos 12 meses. 📉
✔ Se eliminan las variables con varianza 0 (además de otras consideraciones). 🚫
✔ Se normalizan las variables numéricas (únicamente para fines didácticos). ⚖️
Receta <- recipe(delitos ~ ., training(splits)) %>%
step_rm(c(id,pliegue), id = "remover") %>%
step_impute_median(all_predictors(), id = "imputación") %>%
step_geodist(lat = lat, lon = long, log = FALSE,
ref_lat = -34.635706, ref_lon = -58.364521, name = "dist_boca", id = "boca") %>%
step_geodist(lat = lat, lon = long, log = FALSE,
ref_lat = -34.545586, ref_lon = -58.449715, name = "dist_river", id = "river") %>%
step_discretize_xgb(long, outcome = "delitos_last_12", id = "xgb discet") %>%
step_nzv(all_predictors(), id = "varianza 0") %>%
step_normalize(all_numeric(), -all_outcomes(), id = "normalize")# A tibble: 7 x 6
number operation type trained skip id
<int> <chr> <chr> <lgl> <lgl> <chr>
1 1 step rm TRUE FALSE remover
2 2 step impute_median TRUE FALSE imputación
3 3 step geodist TRUE FALSE boca
4 4 step geodist TRUE FALSE river
5 5 step discretize_xgb TRUE FALSE xgb discet
6 6 step nzv TRUE FALSE varianza 0
7 7 step normalize TRUE FALSE normalize 👉🏻 Antes de pasar la receta al flujo de modelado, ¿Puedo ver como resultaron las transformaciones en los datos?
📌 Si 🤩, simplemente con la función juice() (en realidad es un atajo para bake(Receta, new_data = train) )
# A tibble: 21,038 x 36
delitos_last_year delitos_last_12 delitos_last_6 delitos_last_3
<dbl> <dbl> <dbl> <dbl>
1 -0.827 -0.796 -0.787 -0.719
2 -0.827 -0.252 -0.222 -0.285
3 -0.595 -0.655 -0.599 -0.574
4 -0.595 -0.494 -0.523 -0.430
5 -0.827 -0.695 -0.599 -0.502
6 -0.362 -0.312 -0.335 -0.357
7 -0.827 -0.695 -0.523 -0.357
8 -0.362 -0.635 -0.599 -0.647
9 -0.362 -0.554 -0.561 -0.574
10 -0.595 -0.192 -0.146 -0.285
# ... with 21,028 more rows📝 Pequeño truco 👉👉 ¿Deseo ver en detalle qué columnas fueron eliminadas o creadas durante el preprocesamiento? Solo debe agregar el parámetro log_changes = TRUE a la función prep().
oper 1 step rm [training]
step_rm (remover):
removed (2): id, pliegue
oper 2 step impute median [training]
step_impute_median (imputación): same number of columns
oper 3 step geodist [training]
step_geodist (boca):
new (1): dist_boca
oper 4 step geodist [training]
step_geodist (river):
new (1): dist_river
oper 5 step discretize xgb [training]
step_discretize_xgb (xgb discet): same number of columns
oper 6 step nzv [training]
step_nzv (varianza 0):
removed (19): hotel_baja, hotel_alta, cine, hogares_paradores, ...
oper 7 step normalize [training]
step_normalize (normalize): same number of columns
The retained training set is ~ 5.7 Mb in memory.Recipe
Inputs:
role #variables
outcome 1
predictor 54
Training data contained 21038 data points and 21036 incomplete rows.
Operations:
Variables removed id, pliegue [trained]
Median imputation for delitos_last_year, delitos_last_12, d... [trained]
Geographical distances from -34.635706 x -58.364521 using lat, long [trained]
Geographical distances from -34.545586 x -58.449715 using lat, long [trained]
Discretizing variables using xgboost long [trained]
Sparse, unbalanced variable filter removed hotel_baja, hotel_alta, cine,... [trained]
Centering and scaling for delitos_last_year, delitos_last_12, d... [trained]🔎 Se observa para cada paso las columnas eliminadas o creadas. Luego del paso 7 se muestra el siguiente mensaje “The retained training set is ~ 5.71 Mb in memory.” 🤔, esto indica el peso 💾 de la data de entrenamiento dentro de la receta.
📌 Para no guardar estos datos y hacer más liviana la receta, simplemente se especifica el argumento retain = FALSE. “Receta %>% prep(log_changes = TRUE, retain = FALSE, verbose = FALSE)”.
🔹 Esto es recomendable siempre y cuando no se apliquen pasos de preprocesamiento que requieran volver a ver los datos de entrenamiento ✅, o si desea reutilizar la receta para construir recetas más avanzadas 🤖.
❗Luego, se crea un workflow, se agrega un modelo y una receta, se ajusta el workflow sobre la partición de entrenamiento. Finalmente se generan predicciones sobre la partición de prueba.
wf_fit <- workflow() %>%
add_model(Modelo) %>%
add_recipe(Receta) %>%
fit(training(splits))
wf_fit %>%
predict(testing(splits)) %>%
head()# A tibble: 6 x 1
.pred
<dbl>
1 0.596
2 2.62
3 1.10
4 2.36
5 2.96
6 1.65 🔎Se evalúan los resultados.
resultados <- last_fit(wf_fit,splits)
collect_metrics(resultados)# A tibble: 2 x 4
.metric .estimator .estimate .config
<chr> <chr> <dbl> <chr>
1 rmse standard 2.26 Preprocessor1_Model1
2 rsq standard 0.774 Preprocessor1_Model1🎖️ Se muestran los resultados junto al dataset original.
# A tibble: 5,261 x 57
delitos .pred .resid id pliegue delitos_last_year
<dbl> <dbl> <dbl> <chr> <int> <dbl>
1 0 0.605 -0.605 esquina_1009 1 0
2 2 2.61 -0.611 esquina_1011 1 1
3 2 1.05 0.950 esquina_1020 1 0
4 3 2.41 0.590 esquina_103 1 2
5 6 2.85 3.15 esquina_1034 1 4
6 2 1.79 0.214 esquina_104 1 3
7 6 4.78 1.22 esquina_1051 1 1
8 1 3.86 -2.86 esquina_1052 1 7
9 5 1.83 3.17 esquina_1058 1 0
10 11 11.9 -0.861 esquina_1061 1 2
# ... with 5,251 more rowsRemuestreo espacial 🌐
El último caso de uso está relacionado con partición de datos que dependen de ubicaciones geográficas 🗺. A través del siguiente ejemplo, se presenta la utilidad e importancia de este tema 🔎. Mediante la función vfold_cv(), se realiza una particion del conjunto de datos en 6 partes.
set.seed(123)
data_crime <- sknifedatar::data_crime_clime %>% filter(pliegue == 13)
splits_crime <- vfold_cv(data_crime, strata = delitos, v = 6)
splits_crime # 6-fold cross-validation using stratification
# A tibble: 6 x 2
splits id
<list> <chr>
1 <split [1685/338]> Fold1
2 <split [1685/338]> Fold2
3 <split [1685/338]> Fold3
4 <split [1686/337]> Fold4
5 <split [1686/337]> Fold5
6 <split [1688/335]> Fold6¿Cómo podemos ver en el mapa las particiones de las esquinas de CABA ? 💡
Se construye la función plot_splits() (tomada de la documentación de spatialsample 📦, aunque agregando algunas mejoras) y se visualizan las 6 particiones en el mapa.
plot_splits <- function(x,y){
analysis(x) %>%
mutate(partition = "train") %>%
bind_rows(assessment(x) %>%
mutate(partition = "test")) %>%
ggplot(aes(long, lat, color = partition)) +
geom_point(alpha = 0.5) +
theme_minimal() +
labs(title = y)
}🔹 Simplemente se aplica la función plot_splits() sobre las particiones mediante map2(), agregando una nueva columna llamada “graficos” (map, map2, pmap,.. 👉🏻👉🏻👉🏻corresponden a la familia de funciones para el desarrollo de programación funcional ordenada en R, este tema será tratado en un futuro post 🙌)
# 6-fold cross-validation using stratification
# A tibble: 6 x 3
splits id graficos
<list> <chr> <list>
1 <split [1685/338]> Fold1 <gg>
2 <split [1685/338]> Fold2 <gg>
3 <split [1685/338]> Fold3 <gg>
4 <split [1686/337]> Fold4 <gg>
5 <split [1686/337]> Fold5 <gg>
6 <split [1688/335]> Fold6 <gg> 🔹 A continuación, mediante la función ggarrange() se puede generar la visualización suministrando únicamente la columna gráficos previamente calculada.
ggarrange(plotlist = splits_crime$graficos, common.legend = TRUE) %>%
annotate_figure(top = text_grob("Esquinas de CABA: Folds aleatorios",
color = "black", face = "bold", size = 16))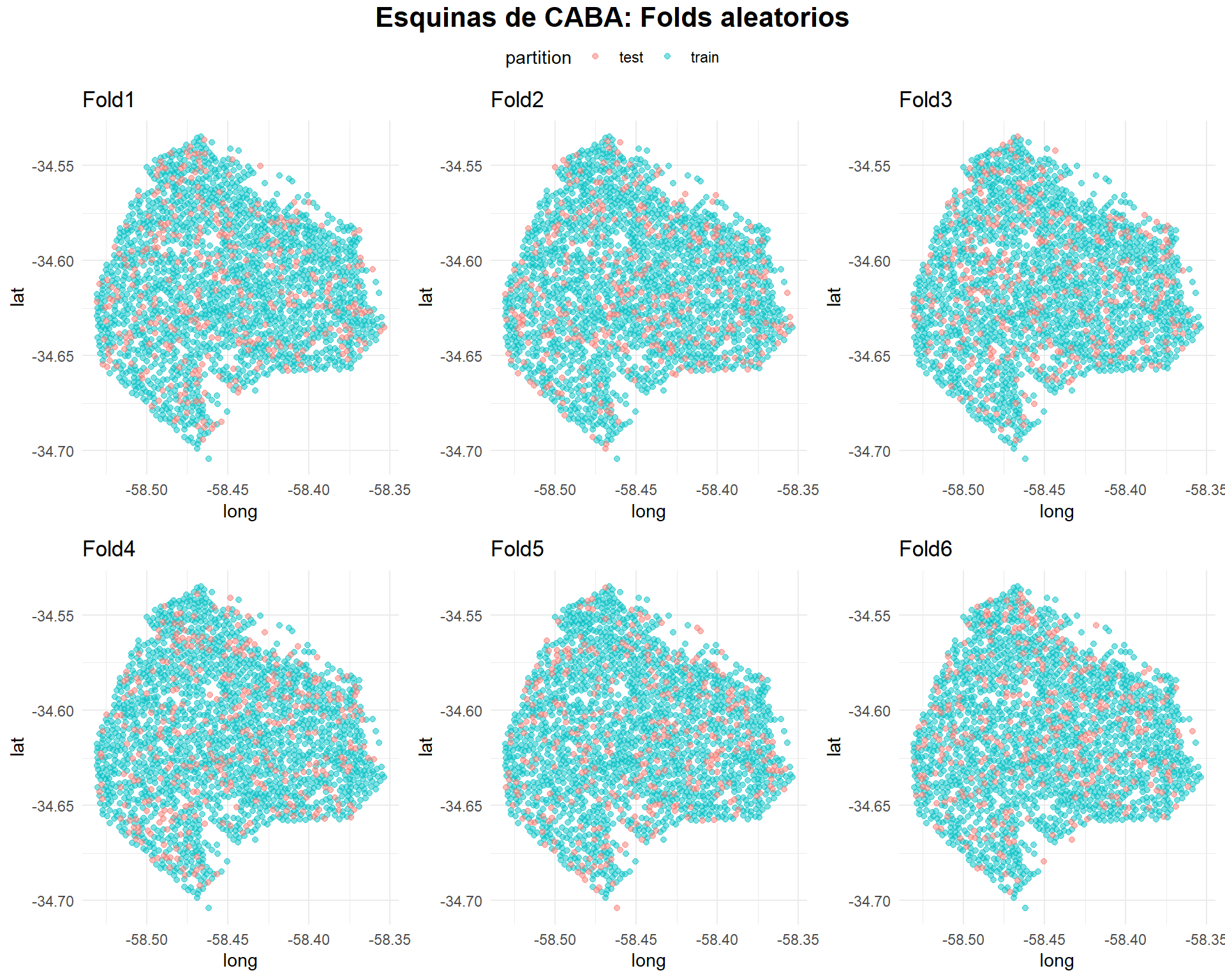
¿ Cuál es el posible problema con estas particiones? 🔎
📌 La evaluación del rendimiento de modelos predictivos de aprendizaje automático, tiene dentro de sus principios 💡 observar el rendimiento de los modelos sobre datos no utilizados durante el entrenamiento. Es decir, 👉🏻 existe un supuesto de independencia entre las observaciones de train y test.
⚠️ Sin embargo, se observa una asignación aleatoria en las particiones de entrenamiento y prueba de cada fold. Al tratarse con datos geoespaciales 🗺, está latente la existencia de autocorrelación espacial en las esquinas de CABA 🔎. Esto violaría el supuesto de independencia mencionado anteriormente 🚨.
💡 Mediante la función spatial_clustering_cv() de spatialsample 📦, puede mitigarse la autocorrelación generando particiones mediante agrupamientos de k-medias. Este es uno de los métodos más sencillos ✅, sin embargo, aún está en desarrollo la integración de técnicas más robustas al ecosistema de tidymodels.
set.seed(123)
splits_crime <- spatial_clustering_cv(data_crime, coords = c("lat", "long"), v = 6)
splits_crime# 6-fold spatial cross-validation
# A tibble: 6 x 2
splits id
<list> <chr>
1 <split [1713/310]> Fold1
2 <split [1712/311]> Fold2
3 <split [1744/279]> Fold3
4 <split [1619/404]> Fold4
5 <split [1653/370]> Fold5
6 <split [1674/349]> Fold6🔹 Siguiendo el caso aleatorio, se aplica la función plot_splits() para observar las particiones en el mapa.
splits_crime <- splits_crime %>% mutate(graficos = map2(splits, id, plot_splits))
ggarrange(plotlist = splits_crime$graficos, common.legend = TRUE) %>%
annotate_figure(
top =text_grob("Esquinas de CABA: Folds K-means",
color = "black", face = "bold", size = 16)
)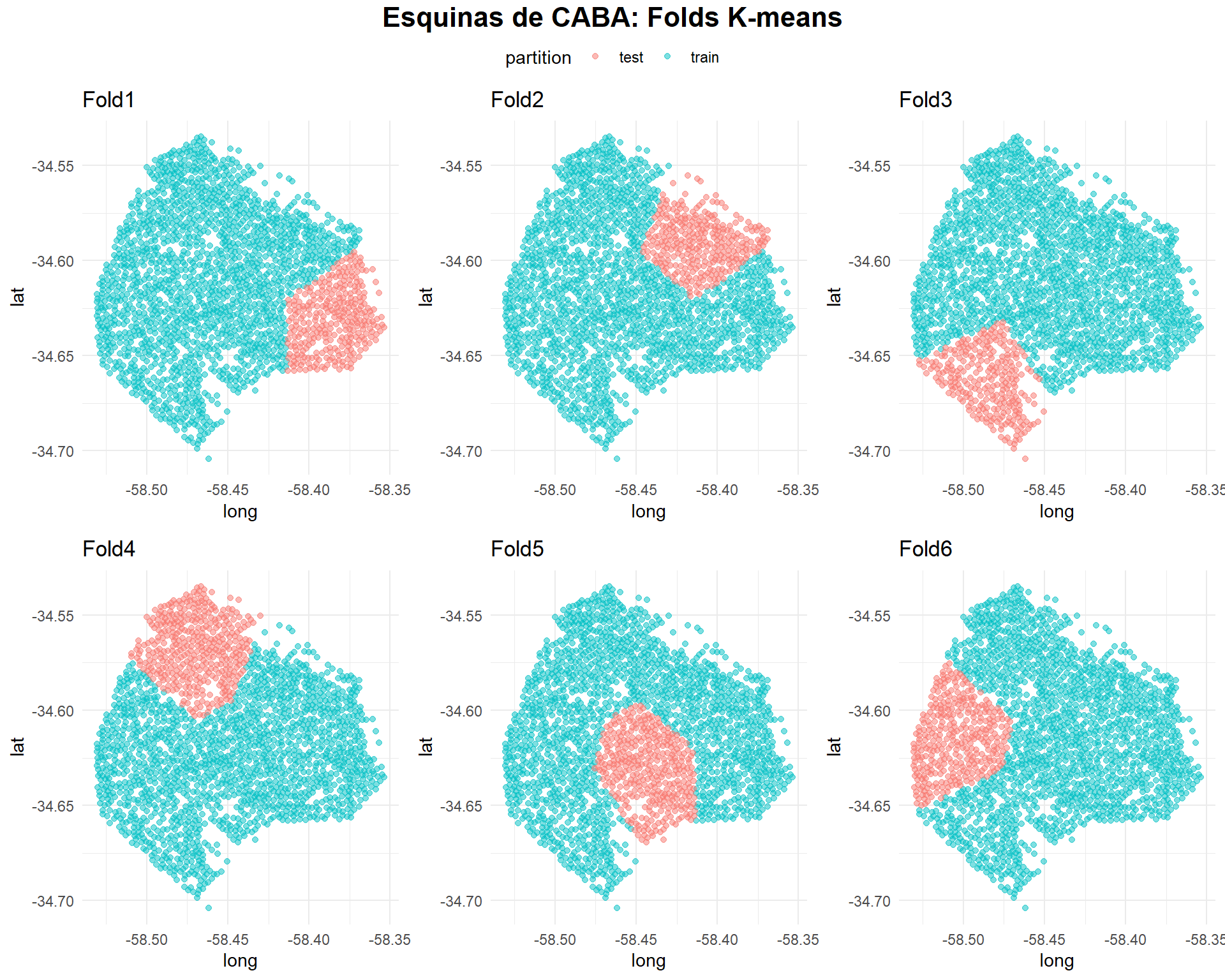
🔹 Se observa un contraste importante entre las 2 particiones. Visualmente, es bastante intuitivo entender las diferencias y aplicaciones de ambos enfoques.
📍 A modo de ejemplo, se evaluará el rendimiento de un modelo de random forest 🤖mediante un workflow 🔁 sobre las 6 particiones. Para esto se creará la función predict_folds() tomada del ejemplo de la documentación de spatialsample 📦, pero con algunas adaptaciones 🛠 para poder incorporar un workflow, la función hace lo siguiente:
✔ Crea un modelo 🤖.
✔ Crea una receta 🍳.
✔ Guarda todo en un workflow y se ajusta sobre la parte de train de un fold 🚀.
✔ Realiza predicciones sobre la parte de test 🔮.
predict_folds <- function(x) {
modelo <- rand_forest() %>%
set_engine("ranger") %>%
set_mode("regression")
receta <- recipe(delitos ~ ., data = analysis(x)) %>%
step_rm(c(id,pliegue), id = "remover")
workflow_crime <- workflow() %>%
add_model(modelo) %>%
add_recipe(receta) %>%
fit(analysis(x))
test <- assessment(x)
tibble::tibble(Longitude = test$long,
Latitude = test$lat,
observed = test$delitos,
predict(workflow_crime, test))
}🔹 Se aplica la función tal sobre cada partición mediante map().
🔹 Se calcula la métrica mae para cada partición.
folds_crime_mae <- folds_crime %>%
unnest(predictions) %>%
group_by(id) %>%
mae(observed, .pred)
folds_crime_mae# A tibble: 6 x 4
id .metric .estimator .estimate
<chr> <chr> <chr> <dbl>
1 Fold1 mae standard 2.05
2 Fold2 mae standard 2.60
3 Fold3 mae standard 1.25
4 Fold4 mae standard 1.33
5 Fold5 mae standard 1.48
6 Fold6 mae standard 1.14🔹 Se visualizan las particiones y su rendimiento de manera conjunta sobre el mapa.
folds_crime %>%
unnest(predictions) %>%
left_join(folds_crime_mae) %>%
ggplot(aes(Longitude, Latitude, color = .estimate)) +
geom_point(alpha = 0.5) +
labs(color = "MAE") +
theme_minimal()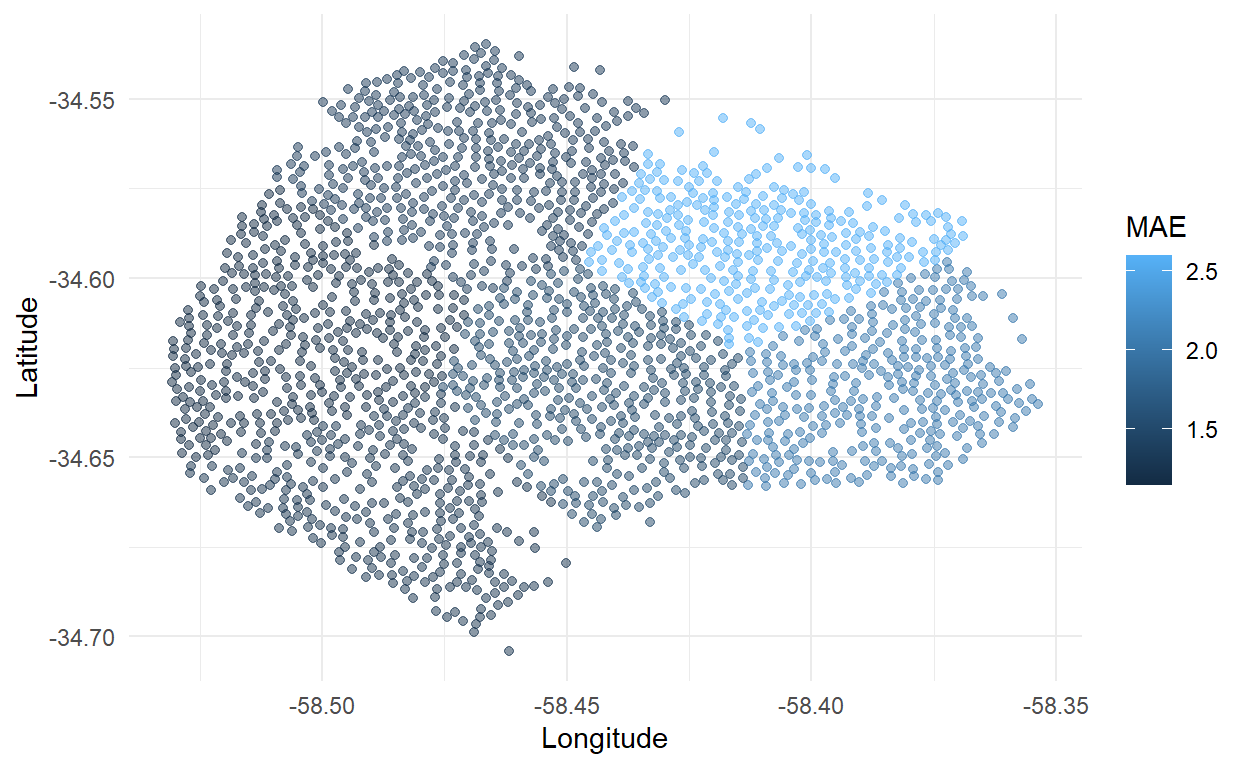
⚠️ No se está realizando un ajuste de hiperparámetros, tampoco un proceso de selección de variables. 👉🏻 Simplemente es un ejemplo didáctico para explicar la utilidad y precauciones que deben tomarse al trabajar con datos geoespaciales 🌐.
Comentarios finales ✍🏻
🔘 El ecosistema tidymodels, representa un cambio disruptivo 🚀 para el análisis de datos en R. La mayoría de los nuevos desarrollos 💡 de modelado predictivo se están desarrollando sobre este ecosistema (series de tiempo 📈, supervivencia 📆, estadística bayesiana 🧪, cluster 💠, SDK para el despliegue en cloud ☁,…).
👉🏻 En esta serie de posts, se intenta explicar (con suerte 🤣) como utilizar tidymodels, mostrando la lógica general de su funcionamiento y posteriormente presentando casos de uso avanzados sobre datos geoespaciales 🌐.
Muchas gracias por leernos 👏🏻👏🏻👏🏻.
Siéntase libre de comentar y compartir 👌🏻.
Contactos ✉
Rafael Zambrano, Linkedin, Twitter, Github, Blogpost.
